本文面向Tanglab及类似的希望针对生物体系做分子动力学模拟的科研人士
本文在一定程度上参考了Amber Tutorials ,本系列教程旨在提供一些作者的使用经验。若想更为全面地掌握Amber分子动力学模拟软件,应当阅读官方教程。
本系列教程所需的预备知识(prerequisite)包括掌握Linux的基本命令,以及对分子动力学模拟理论的大致了解。
关于计算化学的重要性等诸多假大空的套话,此处就不再赘述。从实用性出发,在实际的科研生活中,有相当一部分科研人员需要一定的理论计算来辅助已有的实验成果。倘若将其外包,难免所费不赀;若说合作,不见得有计算组愿意加入进来。倘若自学,又嫌寸金寸阴,唯恐花费太多精力。故笔者编写本系列教程时力求简明,省去所有不必要的理论知识,以期让读者花费最少的时间做出见刊水平(至少HyperChem是不能往论文上放的)的分子动力学模拟。
市面上关于分子动力学软件包实在五花八门。仅就笔者所知的一点浅薄知识出发略作介绍。Gromacs绝对是用户群体最为庞大的软件,其以简便、计算速度快闻名,但同时由于追求运算速度而牺牲了部分功能。Amber尤长于生物体系的分子动力学模拟,功能全面。对于材料体系,则可以考虑使用Lammps。此外VASP也可执行一些AIMD运算。至于与人工智能神经网络等技术结合的分子动力学模拟此处不涉及。总而言之,考虑到本组的研究方向,选择Amber显然是适宜的。
Amber软件包本身实际上分为AmberTools与Amber两部分。前者是免费的,且包含做一个完整的分子动力学模拟的所有功能。Amber是付费的,支持多CPU并行运算与GPU加速。属于锦上添花的内容。贵组服务器上已部署了Amber与AmberTools,可直接使用。或考虑在个人电脑上安装AmberTools。请参考Download Amber与Installation。
相信读者们接触过HyperChem、Chem3D等计算软件,它们的显著特点是all-in-one,即整个体系的建模、参数设置、模拟计算、数据分析等工作全部在一个软件(窗口)内就可以解决。而Gaussian则不是all-in-one的,其由两个软件组成:运算部分为Gaussian,而建模则依赖GaussianView。Amber的架构属于后者,且划分更为细致。Amber实际上只是一系列软件包的一个总称,然而执行具体功能则需要使用不同的软件(类似于office全家桶与word、ppt、excel等的关系)。
先放图。
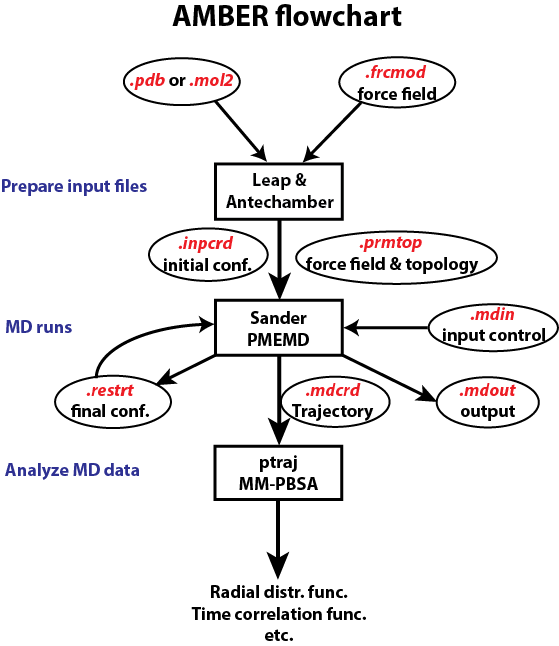 Amber Flowchart
Amber Flowchart
这种图片属于“懂得都懂,不懂我说了也没用”的类型。这里只是先放出来让大家有个初步印象。不过多解释。
总的说来,一个完整的模拟流程包括:
省略号后面列出的是(可能)所需使用的软件。
既然Amber是基于Linux的程序,那么其没有图形化界面[1]是理所应当的了。使用Linux时应抛弃windows的习惯,即找到图标,双击,然后对着窗口点一点就实现功能。直白得讲,使用Amber软件不需要鼠标。该软件也不会有任何图案之类的东西来让你“看得见”它。所有功能都依赖命令行实现。
或许读者期望更为翔实的资源
作为第0篇教程,其作用上只是对AMBER软件包做一个轮廓上的刻画。写到这里就足够了。此外笔者也不喜欢写太长的教程。写着头晕眼花,看着也是如此。
[1] 或许有人会反驳,xleap是有图形化界面的。那确实如此。不过xleap设计得太过闹弹,且在实际使用中其完全可以用命令行程序tleap代替。故本系列教程不会讲解有关xleap的内容。